-
抗生素是治疗和预防细菌感染的重要药物,在人类医疗和动物养殖中使用量大,也是各类废水中常见的污染物[1];医院废水、禽畜养殖废水、抗生素生产废水等都是典型的含抗生素的废水[2]。中国是抗生素生产与出口大国[3],每年生产超过70种大宗抗生素总计约1.3×106 t[4],大宗抗生素原料药的微生物发酵生产过程会产生大量高浓度抗生素残留的废水,其抗生素浓度比其他类别的废水高几个数量级[5],是环境中的高强度抗生素污染源[5]。抗生素生产废水通常采用生化处理方法,高浓度的抗生素抑制微生物的生长,导致生化处理系统去除有机物等污染物的功能恶化[6-7];另外,废水中的抗生素选择压力能够促进废水生物处理细菌群落中抗生素耐药细菌 (antibiotic resistance bacteria,ARB) 和抗生素耐药基因 (antibiotic resistance genes,ARGs) 的富集和传播[8-10],造成潜在的健康危害。因此,选择合适技术对抗生素废水进行处理,对控制抗生素与耐药基因的环境排放至关重要。
由于含有高浓度有机物,抗生素生产废水的处理主要采用厌氧生物处理技术[11-13]。采用两级升流式厌氧污泥床反应器 (up-flow anaerobic sludge bed,UASB) 处理稀释后的发酵类抗生素废水,化学需氧量 (chemical oxygen demand,COD) 总去除率可达到78%~85%[14];采用颗粒污泥膨胀床反应器 (expanded granular sludge blanket,EGSB) 处理阿莫西林废水,可去除85%的COD和80%的阿莫西林[15]。然而,由于高浓度抗生素残留和高含固等水质特征,传统的基于颗粒污泥的厌氧生物处理工艺在处理抗生素废水时存在启动时间长、功能微生物受抗生素扰动、污泥易流失等问题[16],YI等[17]开展UASB处理土霉素生产废水的中试研究,发现土霉素的存在导致有机负荷提升困难、COD去除效率低,在有机负荷为 1.2±0.2 kg·m−3·d−1 (以COD计) 时COD去除率仅为51%±4%。更为重要的是,高浓度抗生素压力下废水生物系统富集的耐药菌和耐药基因会随出水和剩余污泥进入环境[18]。厌氧膜生物反应器 (anaerobic membrane bioreactor,AnMBR) 作为一种新型废水生物处理技术,将厌氧反应器与膜组件进行耦合,具备反应器体积小、污泥产率低、有机负荷高等优势[19],适合处理高含固和高浓度有机废水[20],在餐厨垃圾[21]、城市废水[22]、垃圾渗滤液[23]的处理中展现出良好的性能,在抗生素废水的生物处理方面也展现出良好的应用前景[24-25]。然而,抗生素废水中残留的抗生素不仅会对厌氧生物处理过程产生抑制作用,还会诱导耐药基因在污水、污泥中的赋存。目前已有文献总结了抗生素对厌氧生物过程产生的负面影响[16, 26-27],但其对AnMBR的影响机制和相关应对措施尚未得到全面分析,AnMBR在细菌耐药性风险控制方面的优势与潜力也未见系统报道。
本文在系统总结了AnMBR处理抗生素废水的研究进展,综述了AnMBR在抗生素压力存在下效能变化与耐药基因赋存情况,并对AnMBR处理高浓度抗生素生产废水,实现废水中常规污染物、抗生素与耐药基因协同控制等方面提出展望,以期解决制药废水高效安全处理的行业难题,为我国耐药性风险管控和制药行业的绿色健康发展提供参考。
-
抗生素废水成分复杂,不同药物种类和生产工艺的抗生素废水之间成分差异很大[13]。其中,发酵类抗生素废水通常具有高COD、高氨氮、高悬浮物、高抗生素残留等特征[28-29],化学合成类抗生素废水则通常是具有高生物抑制的难降解高盐废水[30]。很多类型的抗生素废水具有污染物浓度高、难降解且具有生物毒性的特点[31-32]。AnMBR本身的反应器构造使其具备处理抗生素废水的优势。膜组件的存在实现了固体停留时间 (solid retention time,SRT) 和水力停留时间 (hydrolysis retention time,HRT) 的解耦,使反应器对常规污染物和抗生素的去除效果增加;同时通过膜组件将抗生素和携带耐药基因的细菌拦截在反应器内,实现出水中耐药基因的有效削减[33]。
-
由于AnMBR同时具备厌氧技术和膜分离的优点,已有许多研究关注AnMBR对抗生素废水的处理效果 (表1) 。在这些研究中,AnMBR的处理效果差异显著,主要受废水类型、抗生素的种类、抗生素的初始浓度以及反应器的运行参数等的影响。
从表1可以发现,AnMBR处理抗生素废水的有机负荷在1.3~13 kg·m−3·d−1,COD去除率在42.5%到97.0%之间。与UASB、EGSB等传统厌氧废水处理工艺相比,AnMBR所能承受的有机负荷更高,且在抗生素压力下实现更好COD的去除效果[34],这是因为较长的SRT促进了生长较慢的微生物的富集[12],这些微生物的代谢活动实现了对有机物等污染物的高效降解[35-37]。在厌氧发酵的过程中,发酵微生物、共生微生物和产甲烷微生物构成了主要的功能微生物群落[38],这些微生物的协同和共生关系对甲烷发酵过程起着重要作用。相较于传统的厌氧悬浮污泥,厌氧生物膜群落拥有更高丰度的产甲烷微生物[39],因此具有更高的有机物去除率和甲烷产量。
AnMBR对模拟配水中的低浓度抗生素有较高的去除率,而对于基质复杂且抗生素浓度高的实际废水,去除率在34.6%到79.8%之间 (表1) 。厌氧微生物的生物降解功能可能是AnMBR能够去除抗生素的重要原因[40]。AnMBR内高效的产甲烷过程同时是多种抗生素生物转化的关键驱动因素[41-42],反应器中存在的胞外产电菌、互营杆菌、硫酸盐还原菌等特异性微生物对抗生素的降解也有帮助作用[39, 43-46]。除生物降解之外,AnMBR中的膜组件和膜污染层的吸附与截留是实现抗生素削减的重要途经[47-49]。与亲水性抗生素相比,疏水性抗生素更容易被反应器内的污泥相吸附并截留,WIJEKOON等[50]在AnMBR的污泥中发现了阿米替林等疏水性抗生素的积累。然而,目前研究多关注抗生素浓度在 mg·L−1以下的抗生素废水,并主要使用实验室模拟配水,AnMBR处理高浓度抗生素残留的发酵类抗生素生产废水仍需更多的研究。
-
目前针对AnMBR削减耐药基因的研究十分有限,但已有研究证实了膜组件在去除出水中耐药菌和耐药基因上发挥着重要作用。例如,与常规活性污泥处理相比,采用膜生物反应器可以更有效地去除城市污水中的耐药菌,降低耐药基因的丰度[60]。膜孔径以及细胞胶体与膜组件之间的静电相互作用是影响膜吸附截留去除水中耐药基因的重要因素[61-62]。适当的膜污染可以使膜的有效孔径变小,有助于截留抗生素和耐药基因[62]。胞外聚合物 (extracellular polymeric substance,EPS) 和可溶性微生物产物 (soluble microbial product,SMP) 作为膜污染层的主要成分,因其带电官能团和交联结构特性,可以与废水中的耐药基因相互作用并将其拦截在膜上[63-65],实现出水中耐药基因的削减[62, 66]。 因此,AnMBR也可通过膜组件的过滤作用将细菌拦截在反应器内,有效阻止耐药菌与耐药基因进入反应器出水中。WANG等[67]采用AnMBR处理左氧氟沙星废水,发现污染的生物膜可以有效控制耐药基因 (qnrS和qnrA) 的传播。然而,膜污染仍然是阻碍膜生物反应器广泛应用的主要瓶颈问题[68],缓解膜污染与控制耐药基因控制之间的平衡需要进一步研究。
-
对于含低浓度抗生素的废水,AnMBR相比传统厌氧反应器具备更好的污染物去除效果;同时,AnMBR可以通过膜截留控制出水中的抗生素和耐药基因。然而,AnMBR和其他的处理技术对废水中抗生素都存在一定的耐受限值,当水中抗生素的浓度过高时,AnMBR也难以实现高效稳定运行。高抗生素压力会妨碍AnMBR对抗生素废水中的污染物去除,同时导致污泥的细菌耐药性风险问题。
-
抗生素对厌氧消化过程存在多种抑制作用,例如有机酸积累、抑制沼气产生、微生物群落失衡等[69-70],AnMBR在处理抗生素废水的过程中同样存在以上问题。与传统厌氧处理技术相比,AnMBR中高丰度和高多样性的细菌群落保障了高效的厌氧生物降解和产甲烷过程,但由于抗生素对微生物存在抑制和杀灭作用,从而选择性干扰反应器内不同的微生物群,改变微生物物种的相对丰度,影响不同物种之间的相互作用[71],可能会导致AnMBR系统的处理效能降低。然而,目前针对抗生素对AnMBR影响的研究还相对较少。
表2总结了常见的抗生素对厌氧消化过程的抑制研究进展,研究表明不同种类和不同浓度的抗生素对厌氧系统产生的抑制程度不同。而针对同一种抗生素,不同的基质类型、操作条件以及微生物组成也会对其厌氧抑制程度产生较大影响。抗生素的存在会影响细菌对挥发性脂肪酸 (volatile fatty acids,VFAs) 的利用。当微生物群落暴露在高浓度四环素中时,产生VFAs的细菌丰度显著增加,导致挥发性脂肪酸的积累,进而抑制生物产甲烷过程[72-73]。抗生素与VFAs降解菌之间也被发现存在负相关关系[74]。抗生素还可以通过使氢营养型产甲烷菌的占比增加来影响产甲烷菌的群落结构,进而影响甲烷产量[72, 75]。
废水中抗生素浓度的增加同样会改变AnMBR内微生物群落的组成,导致反应器处理效能下降。与AnMBR处理不含抗生素的废水相比,在反应器内添加100 μg·L−1的磺胺类抗生素后,在2周内COD去除率从98.7%±0.91%下降至92.0%±0.08%[86];WEI等[87]发现当AnMBR内的磺胺甲恶唑浓度在10 μg·L−1到1 000 μg·L−1时,COD去除率保持在96%以上不受影响,但磺胺甲恶唑浓度升高至100 mg·L−1时,COD去除率下降至86.2%;使用AnMBR处理环丙沙星废水时发现,4.7 mg·L−1环丙沙星影响微生物物种的系统发育结构、丰富度与多样性,导致COD去除率和甲烷产量等运行参数的下降[88]。
抗生素还会加速膜组件的污染情况,降低膜的使用寿命和处理效能。当反应器受到抗生素等毒性物质冲击时,内部絮体容易发生破损,粒径减小,加速对膜的污染[89-90]。在好氧膜生物反应器中,1 μg·L−1的卡马西平会导致附着在膜上的污泥絮体的平均粒径从85 μm减小至75 μm[91]。抗生素同样也会对AnMBR内部污泥絮体的粒径产生影响,但需要更多直接证据支撑。此外,抗生素的存在会使AnMBR内的厚壁菌门、拟杆菌门和变形菌门等微生物的占比增加[11, 92],这些微生物优先生长在膜的表面,且与污泥中胞外聚合物的生成呈正相关[11],这也说明了抗生素会加剧AnMBR的膜污染过程。
-
采用AnMBR处理含抗生素的废水,由于膜截留功能,出水中耐药基因传播风险得到有效控制,但反应器污泥中仍可能产生和存在耐药基因[93]。AnMBR具有较高的污泥浓度和SRT,可以通过将抗生素吸附到污泥相中减少出水中的抗生素含量[40],携带耐药基因的细菌也会通过膜分离过程留在AnMBR中[94]。采用AnMBR处理含耐药基因的液态食物垃圾,发现该方法实现了出水中耐药基因的去除,但膜上的生物质主要保留或积累了耐药基因[95]。随着废水中磺胺甲恶唑浓度的升高,AnMBR中不动杆菌的数量增加,该结果与不动杆菌被报道为磺胺耐药基因的宿主一致[42]。有研究发现在模拟生活污水中添加10 μg·L−1的红霉素时,AnMBR系统生物质中耐药基因ermF和sul1的丰度显著增加,随着抗生素浓度增加,生物质中的大多数耐药基因都呈增加趋势,这可能与微生物群落结构和耐药基因对生物膜基质的亲和力有关[54]。随着我国对污泥资源化利用的需求日益增长,污泥的资源化处置过程中对耐药基因的控制受到广泛关注[96]。因此,需要在废水进入反应器之前将抗生素去除,从源头阻断耐药基因的传播[97]。
-
AnMBR使传统厌氧处理中存在的污染物去除效率低、污泥易流失、反应器出水传播耐药菌和耐药基因等问题得到有效缓解,但在处理含高浓度抗生素的废水方面仍然存在挑战,污泥中耐药基因富集也需要关注。为此,首先需要构建“抗生素预处理技术+AnMBR”的组合处理工艺,废水中的抗生素在进入AnMBR之前就将其选择性去除;二是可以对AnMBR反应器的结构或工艺进行优化和改造,以进一步提高处理效能和改善膜污染。
-
由于抗生素影响AnMBR的生物效能及可能造成污泥的耐药性风险,因此在抗生素废水进入反应器之前采用预处理手段选择性去除抗生素可以保障生化处理效果,也是控制细菌耐药性发展的最佳途径。常见的预处理手段包括臭氧氧化、碱处理和强化水解技术等[98-99]。
笔者团队前期针对四环素类、β-内酰胺类等具有易水解特性的部分抗生素,开发了选择性去除抗生素残留的强化水解技术,在废水进入生物处理系统之前通过调节pH、温度和加入固体碱催化剂等,可以降低抗生素水解半衰期,实现了抗生素及其效价 (抑菌活性) 的高效去除和耐药基因源头控制[17, 100-102]。HE等[103]通过强化水解技术处理含土霉素废水,实现了土霉素99.7%的去除。通过将该技术与传统厌氧生物处理进行耦合,YI等[17]建立了“强化水解-UASB反应器”的中试装置,发现强化水解技术可消除土霉素对厌氧生化处理阶段的抑制,土霉素效价去除率大于99.9%,厌氧进水负荷从1.5 kg·m−3·d−1提升至接近6 kg·m−3·d−1,COD去除率可达到83.2%,UASB系统污泥总的四环素 (tet) 耐药基因相对丰度和没有采用预处理的UASB相比也减少了60%。进一步,将强化水解预处理应用于河北省某制药厂的土霉素生产废水处理工艺的升级改造中,处理水量为420 m3·d−1,废水中土霉素的浓度为727~1 001 mg·L−1,COD为9 800~12 000 mg·L−1。工程实施后土霉素效价去除率为99%以上,厌氧处理系统的COD去除率为60-70%,和没有预处理的厌氧系统相比,耐药基因降低了83%以上,从而实现了土霉素生产废水中抗生素、耐药基因和常规指标的协同控制[104-106],改造后的吨水处理成本为10~20元,该成本会根据企业现场的热源等条件有所变化。该技术的工程应用被世界卫生组织推荐为制药废水高效处理与耐药性风险控制的典型案例[97]。近期,田野[107]通过小试实验初步探究了“强化水解+AnMBR”应用于土霉素生产废水的可行性,发现AnMBR在有机负荷10 kg·m−3·d−1的条件下能够稳定运行,COD去除率可达到65.0%±1.0%,为AnMBR应用于抗生素生产废水的工程应用提供了重要的基础。
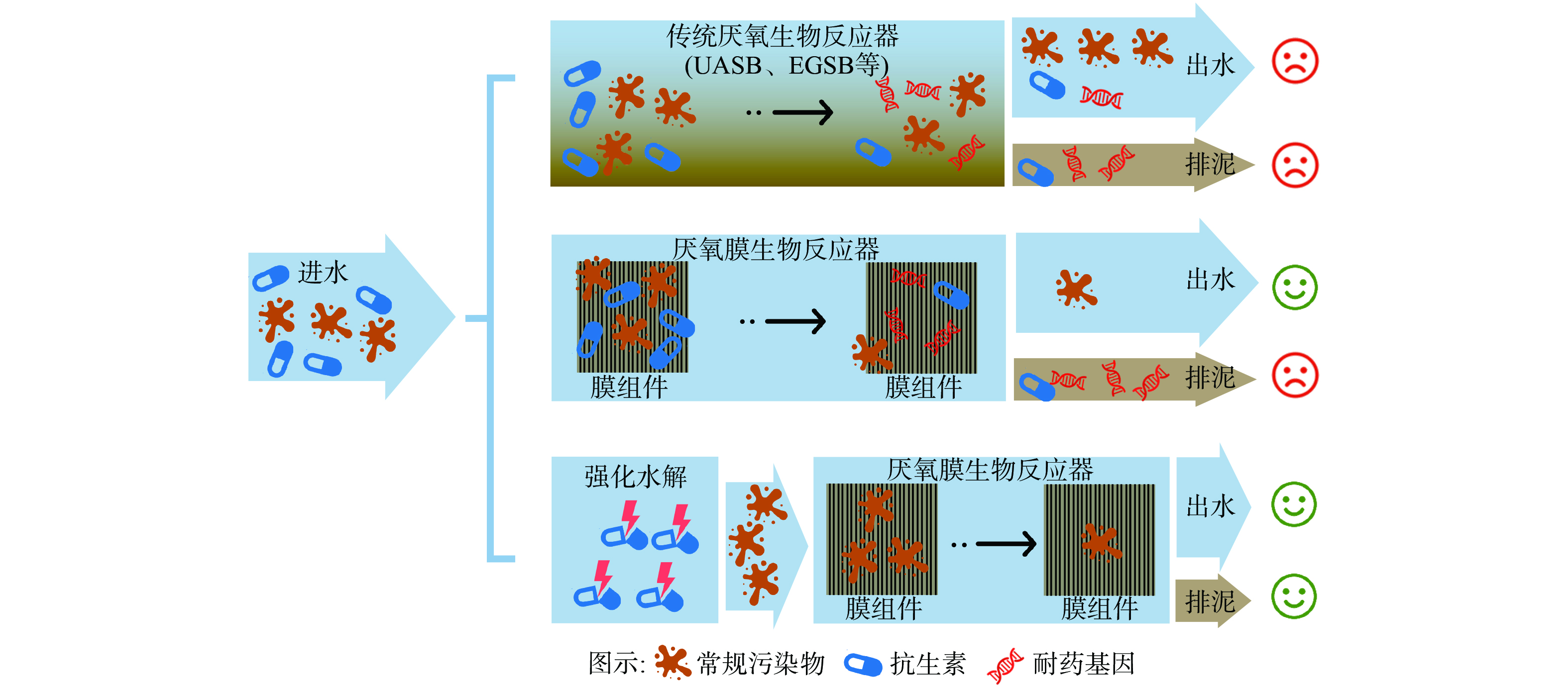
图1总结了各类厌氧生物处理对抗生素废水污染物去除和耐药性削减的效果,传统的厌氧生物反应器在处理高浓度抗生素废水的过程中不仅难以保障出水中污染物的达标排放,也难以避免耐药基因的传播问题;厌氧膜生物反应器可实现对污染物的高效处理,但效能也受抗生素残留浓度的影响。同时,反应器内部的污泥中会有耐药基因产生和富集。因此,在AnMBR前端增加强化水解预处理步骤,消除抗生素对生物处理工艺的负面影响,在实现常规污染物高效去除的同时,为抗生素耐药性控制提供“抗生素水解”和“耐药基因膜截留”的双重保障,有助于实现抗生素废水的高效处理与细菌耐药性风险管控。
最近的研究表明,抗生素在预处理过程中产生的转化产物可能具有与其母体化合物相似的结构,因此也可能贡献效价。畜禽粪便和有机肥中四环素类抗生素的部分转化产物存在一定的效价,尽管其效价弱于母体化合物,但仍然可能促进耐药基因的富集传播[108]。笔者团队的前期工作对红霉素生产废水中红霉素A及其相关物质进行效价分析,发现除母体外,红霉素A的2种生产副产物和3种转化产物对废水中的抗生素效价同样具有重要贡献[78]。土霉素的两种主要水解产物缺乏抑菌活性,但可能是土霉素废水中不可生物降解有机物的重要组成部分[83],可在“强化水解+AnMBR”工艺后进一步增加深度处理工艺,保障末端水质。
-
在废水中的抗生素被选择性去除之后,为了进一步实现出水水质的提升,并减少膜污染,可以通过将现有的AnMBR与高温厌氧、电化学等技术耦合,进行厌氧膜生物反应器工艺优化。常见的优化AnMBR有高温厌氧膜生物反应器、厌氧流化床膜生物反应器和厌氧电化学膜生物反应器等。其中,高温厌氧膜生物反应器在一定的水质和工程条件下具有很好的应用前景,其结合了嗜热生物处理的优势,提高了生物降解的速度,同时由于膜的存在,解决了嗜热细菌絮凝和沉降能力差的问题,保持较高的污泥浓度。升温有利于抗生素的水解,高温体系中的嗜热微生物群落也能够进一步促进抗生素的生物降解[109-111]。高温条件也有助于消除耐药基因和其宿主菌,例如利用高温厌氧反应器处理含高浓度土霉素的模拟废水,污泥中的耐药基因宿主菌丰度占比从41.74%±2.60%下降至12.08%±1.02%[112]。高温厌氧膜生物反应器适合处理高有机负荷的含热废水,或前端经过高温预处理的有机废水。由于热处理会影响聚合物膜的稳定性和耐久性,而热稳定性更高的无机膜具有更高的成本[109],开发热稳定性和耐久性更好的膜材料是高温厌氧膜生物反应器发展的研究方向之一。
厌氧流化床膜生物反应器是在AnMBR的基础上在反应器中投加生物膜载体 (如活性炭、聚合氯化铝、海绵等) 而形成的新型工艺,在减缓膜污染方面具有优势。生物膜载体的投加能够提升污泥颗粒的粒径、絮凝性和沉降性,减少水中胞外聚合物的浓度,并促进抗生素降解菌的生长和富集[11]。在膜生物反应器中添加载体,一方面可以通过载体的吸附作用去除胶体物质,降低反应器内EPS和SMP等物质的浓度来减缓膜污染的形成,另一方面可以通过对膜的冲刷作用减少膜污染[113]。采用添加海绵的膜生物反应器处理含抗生素的医院废水,对诺氟沙星、氧氟沙星、环丙沙星、四环素、甲氧苄啶等抗生素具有显著的去除效果[114]。但反应器长期运行过程中,载体的吸附能力逐渐耗尽并可能发生破裂,反而会加速对膜的污染[115]。因此在实际应用的过程中,载体的维护与更换是亟需解决的问题。
在膜生物反应器中引入适宜强度的电场可以增强微生物活性,增加污泥表面电荷,从而使膜表面污泥层的稳定性降低,有效缓解膜污染问题。采用厌氧电化学膜生物反应器处理含氯四环素废水,高效去除高浓度氯四环素 (45 mg·L−1) 的同时减少出水中耐药基因的增殖[116]。但施加外部电压后,发现污泥中四环素降解基因tetX和磺胺失活基因 (mphA-01和drfA12) 富集,外加电场可能导致耐药基因水平转移和微生物群落的转移[117]。电压大小对反应器膜污染控制和ARGs变化的影响还需要进一步研究。DING等[118]研究认为0.6 V为兼顾性能和经济性的最佳电压,高压条件 (大于0.8 V) 会加重膜污染,抑制COD去除率和产甲烷活性。目前关于优化AnMBR处理抗生素废水的研究还处在可行性的实验室探索阶段,特别是对于厌氧流化床膜生物反应器和厌氧电化学膜生物反应器的工程应用还需要进一步探索,对上述优化和改进的膜反应器处理抗生素废水的运行机制、处理效果和影响因素等也需要进行更深入的研究。
-
1) AnMBR在处理抗生素废水方面展现出良好的应用前景,通过将膜组件与厌氧反应器耦合,实现反应器内厌氧微生物的富集,促进了废水中难降解有机物和抗生素等的降解,提升出水品质和甲烷产量,同时通过膜组件也可将抗生素和携带耐药基因的细菌拦截在反应器内。
2) 抗生素会影响AnMBR中微生物的厌氧处理过程和群落结构,导致处理性能降低并造成污泥中抗生素和耐药基因的累积。抗生素的种类和浓度、废水组成、处理工艺的不同都会导致抗生素对厌氧处理影响程度的差异。抗生素的存在会加速反应器的膜污染状况。
3) 在AnMBR之前将废水中的抗生素效价去除是保障抗生素废水常规污染物、抗生素和耐药基因协同消减的最佳策略,为此提出针对抗生素废水的“抗生素效价选择性去除预处理-AnMBR”组合工艺,可利用强化水解等预处理技术实去除进水中的抗生素效价残留,也能够有效消减污泥中的抗生素和耐药基因,实现废水的高效短流程处理。
目前AnMBR处理抗生素废水的相关研究仍然比较有限,综合国内外含抗生素废水处理的基础研究和工程应用的发展情况,而未来可以从以下两个方面开展研究。
1) 抗生素及其转化产物对AnMBR处理效能和细菌耐药性发展的影响。未来的研究需要关注更多的抗生素类型,对不同抗生素水解和氧化等特性开展更深入的研究,由此选择合理高效的预处理和末端保障技术。此外,部分抗生素的转化产物具有一定的效价,或对难降解COD具有贡献。因此,一方面需要关注抗生素及转化产物对细菌耐药性发展的影响和贡献;另一方面继续进行高效预处理和末端保障技术的研发,促进抗生素向低耐药性、易生物降解产物的定向转化。
2) 厌氧膜生物反应器处理高浓度抗生素废水的基础研究和工程探索。加强对AnMBR处理高浓度抗生素残留的发酵类抗生素废水的研究,以及“抗生素效价去除预处理+AnMBR”等工艺的技术优化和在实际废水中的应用研究。对新型厌氧膜生物反应器处理含抗生素废水的运行效果、膜污染控制和影响因素开展探究,通过现场中试和工程应用推进AnMBR在制药等行业抗生素废水处理的工程化。
厌氧膜生物反应器处理抗生素废水研究进展与展望
Research progress and prospect of anaerobic membrane bioreactor for treatment of antibiotic wastewater
-
摘要: 中国是抗生素生产大国,抗生素生产过程伴随产生大量的含抗生素残留的有机废水,通常采用厌氧生物技术进行处理。然而传统的厌氧处理技术对抗生素废水存在效能不高的问题,并且难以实现废水中常规污染物、抗生素与耐药基因的协同控制。厌氧膜生物反应器同时具有厌氧处理与膜处理技术的优点,在处理抗生素废水方面展现出很好的应用前景。本文总结了厌氧膜生物反应器处理抗生素废水的研究进展,从常规污染物去除和耐药基因削减两方面阐述了厌氧膜生物反应器的处理优势;重点梳理了抗生素对厌氧膜生物反应处理过程中生物效能的抑制和耐药基因赋存的影响。在此基础上,提出“强化水解预处理去除抗生素残留效价 (抑菌活性) -厌氧膜生物反应器”组合处理工艺作为短流程的抗生素废水处理最佳策略,在提升污水处理效能的同时实现对耐药性的协同控制,为制药废水绿色、高效和安全处理提供参考。Abstract: China is a major producer of antibiotics in the world, and the production process of antibiotics is accompanied by a large amount of wastewater containing high concentration of antibiotics. However, it is difficult to simultaneously remove conventional pollutants, antibiotics, and antibiotic resistance genes from antibiotic production wastewater by traditional anaerobic biological treatment technology. Anaerobic membrane bioreactor (AnMBR) combines the advantages of anaerobic treatment and membrane technology, and exhibits potential for treating antibiotics-containing wastewater. Based on the literature review, this paper summarized the current progress of antibiotics wastewater treatment using AnMBR, which showed the advantages on pollutant removal and reduction of antibiotic resistance genes. The impacts of antibiotics on microbial inhibition and enrichment of antibiotic resistance genes were focused. Moreover, this study proposed that technique integration of “enhanced hydrolysis pretreatment to remove antibiotic -AnMBR” was optimal short-flow treatment approach for antibiotic production wastewater. This technique integration could simultaneously improve wastewater treatment efficiency and antibiotic resistance control, which is the reference for green, efficient and safe treatment of pharmaceutical wastewater.
-
-
表 1 厌氧膜生物反应器处理抗生素废水的研究进展
Table 1. Research progress of AnMBR treating antibiotic-containing wastewater
抗生素类型 废水类型 HRT/h OLR/
(kg·m−3·d−1)MLSS/
(g·L−1)进水COD/
(mg·L−1)COD
去除率/%初始抗生素
质量浓度抗生素
去除率/%参考文献 阿莫西林 实际废水 48.1~23.9 2.4~4.5 16.7±0.5 3 601~5 919 90.3±1.5 3.8~10.9 mg·L−1 73.2±4.3 [51] 头孢曲松 2.9~5.7 mg·L−1 47.7±2.2 头孢哌酮 0.4~1.0 mg·L−1 79.4±4.1 氨苄西林 4.8~9.5 mg·L−1 34.6±3.3 青霉素 实际废水 30.6 13.0±0.6 9.5~10.2 16 249±714 60.3±2.8 — — [52] 青霉素 实际废水 42.6 7.7~9.2 11.0 15 365±1 214 42.5±4.3 — — [53] 阿莫西林 实际废水 20.0 — — 1 800~8 000 88.9 19.7~214.7 mg·L−1 79.8 [15] 磺胺甲恶唑 模拟配水 16.0 — 8.2±0.5 400-600 93±3.1 10~250 μg·L−1 71.0~85.0 [54] 红霉素 10~250 μg·L−1 67.0~88.0 氨苄西林 10~250 μg·L−1 94.0~98.0 四氢呋喃 模拟配水 48.0 3.9~4.1 16.5 8 014~22 077 96.7 1 709~3 057 mg·L−1 98.0 [55] 甲氧苄啶 模拟配水 24.0~6.0 2.0 5.8~8.1 500 93.9±1.8 1~4 μg·L−1 94.2±5.5 [56] 磺胺甲恶唑 1~3 μg·L−1 67.8±13.9 磺胺甲恶唑 模拟配水 12.0 1.3 4.9~6.8 400±10 97.0 10~20 μg·L−1 98.0 [57] 苯并噻唑 模拟配水 24.0 3.1 7.2 2 961~3 337 90.9~93.6 50 mg·L−1 97.6±0.5 [58] 磺胺嘧啶 模拟配水 1.3 5.7 30.0 250 95.0 18.9 ng·L−1 93.7 [59] 磺胺甲恶唑 312 ng·L−1 89.1 红霉素 319 ng·L−1 86.3 克拉霉素 324 ng·L−1 89.0 表 2 抗生素对厌氧消化过程的抑制情况
Table 2. The inhibition of antibiotics on anaerobic digestion process
抗生素 基质类型 抑制浓度/
(mg·L−1)抑制情况 参考文献 红霉素 活性污泥 50 抑制厌氧消化的水解过程 [76] 模拟配水 200 挥发性脂肪酸增加 [77] 实际废水 5 金葡萄球菌被红霉素A及其效价 (抑菌活性) 相关物质完全抑制 [78] 磺胺甲恶唑 模拟配水 100 累计甲烷产量降低,500 mg·L−1时完全抑制产甲烷 [79] 剩余污泥 0.24 产气量下降48%,短链脂肪酸是对照组的1.73倍 [80] 四环素 模拟配水 500 完全抑制产甲烷过程 [79] 模拟配水 0.25 沼气总产量下降39.23%,产氢产酸增加 [81] 模拟配水 8.5 厌氧系统崩溃,停止投加四环素也无法恢复 [75] 土霉素 模拟配水 150 UASB反应器崩溃,200mg·L−1时反应器进一步恶化 [82] 模拟配水 500 甲烷产量被抑制,与对照组相比降低64.6% [83] 阿莫西林 污水污泥 1 024 抑制甲烷发酵过程中乙酸生成,沼气中甲烷含量降低 [84] 污水污泥 1 024 使沼气中的甲烷含量降低至44%,显著增加丁酸浓度 [85] -
[1] BENGTSSON-PALME J, LARSSON D G J. Concentrations of antibiotics predicted to select for resistant bacteria: Proposed limits for environmental regulation[J]. Environment International, 2016, 86: 140-149. doi: 10.1016/j.envint.2015.10.015 [2] ZHANG Y, WALSH T R, WANG Y, et al. Minimizing risks of antimicrobial resistance development in the environment from a public one health perspective[J]. China CDC Weekly, 2022, 4(49): 1105-1109. doi: 10.46234/ccdcw2022.224 [3] 李超. 制药产业国际竞争力关键因素与形成机理研究[D]. 北京: 中国政法大学, 2020. [4] HAN Z M, ZHANG Y, YANG M. Deterring the transmission of amr in the environment: A chinese perspective. Handbook on antimicrobial resistance: current status, trends in detection and mitigation measures[EB/OL]. [2023-04-22].https://link.springer.com/referenceworkentry/10.1007/978-981-16-9723-4_52-1. [5] LARSSON D G J, FLACH C F. Antibiotic resistance in the environment[J]. Nature Reviews Microbiology, 2021, 20(5): 257-269. [6] ZHANG Y, XIE J P, LIU M M, et al. Microbial community functional structure in response to antibiotics in pharmaceutical wastewater treatment systems[J]. Water Research, 2013, 47(16): 6298-6308. doi: 10.1016/j.watres.2013.08.003 [7] MA W L, QI R, ZHANG Y, et al. Performance of a successive hydrolysis, denitrification and nitrification system for simultaneous removal of COD and nitrogen from terramycin production wastewater[J]. Biochemical Engineering Journal, 2009, 45(1): 30-34. doi: 10.1016/j.bej.2009.02.001 [8] ZHANG Q Q, YING G G, PAN C G, et al. Comprehensive evaluation of antibiotics emission and fate in the river basins of china: Source analysis, multimedia modeling, and linkage to bacterial resistance[J]. Environmental Science & Technology, 2015, 49(11): 6772-6782. [9] LI D, QI R, YANG M, et al. Bacterial community characteristics under long-term antibiotic selection pressures[J]. Water Research, 2011, 45(18): 6063-6073. doi: 10.1016/j.watres.2011.09.002 [10] 张昱, 冯皓迪, 唐妹, 等. β-内酰胺类抗生素的环境行为与制药行业源头控制技术研究进展[J]. 环境工程学报, 2020, 14(8): 1993-2010. [11] CHENG D L, NGO H H, GUO W S, et al. Anaerobic membrane bioreactors for antibiotic wastewater treatment: Performance and membrane fouling issues[J]. Bioresource Technology, 2018, 267: 714-724. doi: 10.1016/j.biortech.2018.07.133 [12] CHENG D L, NGO H H, GUO W S, et al. Bioprocessing for elimination antibiotics and hormones from swine wastewater[J]. Science of The Total Environment, 2018, 621: 1664-1682. doi: 10.1016/j.scitotenv.2017.10.059 [13] ENIOLA J O, KUMAR R, BARAKAT M A, et al. A review on conventional and advanced hybrid technologies for pharmaceutical wastewater treatment[J]. Journal of Cleaner Production, 2022, 356. [14] 刘玮. 2级厌氧反应器处理发酵类抗生素废水[J]. 水处理技术, 2018, 144(7): 78-81. [15] MENG L W, LI X K, WANG K, et al. Influence of the amoxicillin concentration on organics removal and microbial community structure in an anaerobic egsb reactor treating with antibiotic wastewater[J]. Chemical Engineering Journal, 2015, 274: 94-101. doi: 10.1016/j.cej.2015.03.065 [16] SHI X, LEONG K Y, NG H Y. Anaerobic treatment of pharmaceutical wastewater: A critical review[J]. Bioresource Technology, 2017, 245(Pt A): 1238-1244. [17] YI Q Z, ZHANG Y, GAO Y X, et al. Anaerobic treatment of antibiotic production wastewater pretreated with enhanced hydrolysis: Simultaneous reduction of COD and ARGs[J]. Water Research, 2017, 110: 211-217. doi: 10.1016/j.watres.2016.12.020 [18] JU F, BECK K, YIN X L, et al. Wastewater treatment plant resistomes are shaped by bacterial composition, genetic exchange, and upregulated expression in the effluent microbiomes[J]. The ISME Journal, 2018, 13(2): 346-360. [19] 程辉, 纪佳渊, 李玉友. 厌氧膜生物反应器及其应用研究实例[J]. 生物产业技术, 2019, 2: 15-27. [20] LIAO B Q, KRAEMER J T, BAGLEY D M. Anaerobic membrane bioreactors: Applications and research directions[J]. Critical Reviews in Environmental Science and Technology, 2006, 36(6): 489-530. doi: 10.1080/10643380600678146 [21] CHENG H, HIRO Y, HOJO T, et al. Upgrading methane fermentation of food waste by using a hollow fiber type anaerobic membrane bioreactor[J]. Bioresource Technology, 2018, 267: 386-394. doi: 10.1016/j.biortech.2018.07.045 [22] LIN H J, CHEN J R, WANG F Y, et al. Feasibility evaluation of submerged anaerobic membrane bioreactor for municipal secondary wastewater treatment[J]. Desalination, 2011, 280(1/2/3): 120-126. [23] ABUABDOU S M A, AHMAD W, AUN N C, et al. A review of anaerobic membrane bioreactors (AnMBR) for the treatment of highly contaminated landfill leachate and biogas production: Effectiveness, limitations and future perspectives[J]. Journal of Cleaner Production, 2020, 255: 120215. doi: 10.1016/j.jclepro.2020.120215 [24] LI S N, ZHANG C F, LI F X, et al. Technologies towards antibiotic resistance genes (ARGs) removal from aquatic environment: A critical review[J]. Journal of Hazardous Materials, 2021, 411: 125148. doi: 10.1016/j.jhazmat.2021.125148 [25] OBEROI A S, SURENDRA K C, WU D, et al. Anaerobic membrane bioreactors for pharmaceutical-laden wastewater treatment: A critical review[J]. Bioresource Technology, 2022, 361: 127667. doi: 10.1016/j.biortech.2022.127667 [26] WU Q D, ZOU D S, ZHENG X C, et al. Effects of antibiotics on anaerobic digestion of sewage sludge: Performance of anaerobic digestion and structure of the microbial community[J]. Science of the Total Environment, 2022, 845: 157384. doi: 10.1016/j.scitotenv.2022.157384 [27] CHENG D L, NGO H H, GUO W S, et al. Problematic effects of antibiotics on anaerobic treatment of swine wastewater[J]. Bioresource Technology, 2018, 263: 642-653. doi: 10.1016/j.biortech.2018.05.010 [28] WANG G W, WANG D, XU X C, et al. Wet air oxidation of pretreatment of pharmaceutical wastewater by Cu2+ and [PxWmOy]q- co-catalyst system[J]. Journal of Hazardous Materials, 2012, 217: 366-373. [29] OKTEM Y A, INCE O, SALLIS P, et al. Anaerobic treatment of a chemical synthesis-based pharmaceutical wastewater in a hybrid upflow anaerobic sludge blanket reactor[J]. Bioresource Technology, 2008, 99(5): 1089-1096. doi: 10.1016/j.biortech.2007.02.036 [30] CHEN Z B, XU J, HU D X, et al. Performance and kinetic model of degradation on treating pharmaceutical solvent wastewater at psychrophilic condition by a pilot-scale anaerobic membrane bioreactor[J]. Bioresource Technology, 2018, 269: 319-328. doi: 10.1016/j.biortech.2018.08.075 [31] ZHANG Y, TIAN Z, LIU M M, et al. High concentrations of the antibiotic spiramycin in wastewater lead to high abundance of ammonia-oxidizing archaea in nitrifying populations[J]. Environmental Science & Technology, 2015, 49(15): 9124-32. [32] LUAN X, ZHANG H, TIAN Z, et al. Microbial community functional structure in an aerobic biofilm reactor: Impact of streptomycin and recovery[J]. Chemosphere, 2020, 255: 127032. doi: 10.1016/j.chemosphere.2020.127032 [33] MUNIR M, WONG K, XAGORARAKI I. Release of antibiotic resistant bacteria and genes in the effluent and biosolids of five wastewater utilities in michigan[J]. Water Research, 2011, 45(2): 681-693. doi: 10.1016/j.watres.2010.08.033 [34] RANI J, PANDEY KP, KUSHWAHA J, et al. Antibiotics in anaerobic digestion: Investigative studies on digester performance and microbial diversity[J]. Bioresource Technology, 2022, 361: 127662. doi: 10.1016/j.biortech.2022.127662 [35] VAN LIER J B. High-rate anaerobic wastewater treatment: Diversifying from end-of-the-pipe treatment to resource-oriented conversion techniques[J]. Water Science and Technology, 2008, 57(8): 1137-1148. doi: 10.2166/wst.2008.040 [36] TAO Y, GAO D W, FU Y, et al. Impact of reactor configuration on anammox process start-up: MBR versus SBR[J]. Bioresource Technology, 2012, 104: 73-80. doi: 10.1016/j.biortech.2011.10.052 [37] DERELI R K, ERSAHIN M E, OZGUN H, et al. Potentials of anaerobic membrane bioreactors to overcome treatment limitations induced by industrial wastewaters[J]. Bioresource Technology, 2012, 122: 160-70. doi: 10.1016/j.biortech.2012.05.139 [38] CHENG H, CHENG D, MAO J W, et al. Identification and characterization of core sludge and biofilm microbiota in anaerobic membrane bioreactors[J]. Environment International, 2019, 133: 105165. doi: 10.1016/j.envint.2019.105165 [39] HARB M, ZAREI-BAYGI A, WANG P, et al. Antibiotic transformation in an anaerobic membrane bioreactor linked to membrane biofilm microbial activity[J]. Environmental Research, 2021, 200: 111456. doi: 10.1016/j.envres.2021.111456 [40] MONSALVO V M, MCDONALD J A, KHAN S J, et al. Removal of trace organics by anaerobic membrane bioreactors[J]. Water Research, 2014, 49: 103-112. doi: 10.1016/j.watres.2013.11.026 [41] GONZALEZ-GIL L, MAURICIO-IGLESIAS M, SERRANO D, et al. Role of methanogenesis on the biotransformation of organic micropollutants during anaerobic digestion[J]. Science of the Total Environment, 2018, 622-623: 459-466. doi: 10.1016/j.scitotenv.2017.12.004 [42] CETECIOGLU Z, INCE B, ORHON D, et al. Anaerobic sulfamethoxazole degradation is driven by homoacetogenesis coupled with hydrogenotrophic methanogenesis[J]. Water Research, 2016, 90: 79-89. doi: 10.1016/j.watres.2015.12.013 [43] SAWAYA C B, HARB M. Considering the prospect of utilizing anaerobic membrane biofouling layers advantageously for the removal of emerging contaminants[J]. Frontiers in Chemical Engineering, 2021, 3: 642280. doi: 10.3389/fceng.2021.642280 [44] HARB M, WEI C H, WANG N, et al. Organic micropollutants in aerobic and anaerobic membrane bioreactors: Changes in microbial communities and gene expression[J]. Bioresource Technology, 2016, 218: 882-891. doi: 10.1016/j.biortech.2016.07.036 [45] JIA Y Y, KHANAL S K, ZHANG H Q, et al. Sulfamethoxazole degradation in anaerobic sulfate-reducing bacteria sludge system[J]. Water Research, 2017, 119: 12-20. doi: 10.1016/j.watres.2017.04.040 [46] JIA Y Y, ZHANG H Q, KHANAL S K, et al. Insights into pharmaceuticals removal in an anaerobic sulfate-reducing bacteria sludge system[J]. Water Research, 2019, 161: 191-201. doi: 10.1016/j.watres.2019.06.010 [47] JI J, KAKADE A, YU Z S, et al. Anaerobic membrane bioreactors for treatment of emerging contaminants: A review[J]. Journal of Environmental Management, 2020, 270: 110913. doi: 10.1016/j.jenvman.2020.110913 [48] XIE J, DUAN X, FENG L Y, et al. Influence of sulfadiazine on anaerobic fermentation of waste activated sludge for volatile fatty acids production: Focusing on microbial responses[J]. Chemosphere, 2019, 219: 305-312. doi: 10.1016/j.chemosphere.2018.12.015 [49] ZHENG W L, WEN X H, ZHANG B, et al. Selective effect and elimination of antibiotics in membrane bioreactor of urban wastewater treatment plant[J]. Science of The Total Environment, 2019, 646: 1293-1303. doi: 10.1016/j.scitotenv.2018.07.400 [50] WIJEKOON K C, MCDONALD J A, KHAN S J, et al. Development of a predictive framework to assess the removal of trace organic chemicals by anaerobic membrane bioreactor[J]. Bioresource Technology, 2015, 189: 391-398. doi: 10.1016/j.biortech.2015.04.034 [51] HUANG B, WANG H C, CUI D, et al. Treatment of pharmaceutical wastewater containing β-lactams antibiotics by a pilot-scale anaerobic membrane bioreactor (AnMBR)[J]. Chemical Engineering Journal, 2018, 341: 238-247. doi: 10.1016/j.cej.2018.01.149 [52] NG K K, SHI X Q, NG H Y. Evaluation of system performance and microbial communities of a bioaugmented anaerobic membrane bioreactor treating pharmaceutical wastewater[J]. Water Research, 2015, 81: 311-24. doi: 10.1016/j.watres.2015.05.033 [53] NG K K, SHI X Q, TANG M K Y, et al. A novel application of anaerobic bio-entrapped membrane reactor for the treatment of chemical synthesis-based pharmaceutical wastewater[J]. Separation and Purification Technology, 2014, 132: 634-643. doi: 10.1016/j.seppur.2014.06.021 [54] ZAREI-BAYGI A, HARB M, WANG P, et al. Evaluating antibiotic resistance gene correlations with antibiotic exposure conditions in anaerobic membrane bioreactors[J]. Environmental Science & Technology, 2019, 53(7): 3599-3609. [55] HU D X, SU H Y, CHEN Z B, et al. Performance evaluation and microbial community dynamics in a novel AnMBR for treating antibiotic solvent wastewater[J]. Bioresource Technology, 2017, 243: 218-227. doi: 10.1016/j.biortech.2017.06.095 [56] XIAO Y Y, YAOHARI H, DE ARAUJO C, et al. Removal of selected pharmaceuticals in an anaerobic membrane bioreactor (AnMBR) with/without powdered activated carbon (PAC)[J]. Chemical Engineering Journal, 2017, 321: 335-345. doi: 10.1016/j.cej.2017.03.118 [57] LEIKNES T, AMY G, HOPPE-JONES C, et al. Organic micro-pollutants’ removal via anaerobic membrane bioreactor with ultrafiltration and nanofiltration[J]. Journal of Water Reuse and Desalination, 2016, 6(3): 362-370. doi: 10.2166/wrd.2015.138 [58] LI Y, HU Q, GAO D W. Dynamics of archaeal and bacterial communities in response to variations of hydraulic retention time in an integrated anaerobic fluidized-bed membrane bioreactor treating benzothiazole wastewater[J]. Archaea, 2018, 2018: 9210534. [59] DUTTA K, LEE M Y, LAI W W, et al. Removal of pharmaceuticals and organic matter from municipal wastewater using two-stage anaerobic fluidized membrane bioreactor[J]. Bioresource Technology, 2014, 165: 42-9. doi: 10.1016/j.biortech.2014.03.054 [60] LE T H, NG C, TRAN N H, et al. Removal of antibiotic residues, antibiotic resistant bacteria and antibiotic resistance genes in municipal wastewater by membrane bioreactor systems[J]. Water Research, 2018, 145: 498-508. doi: 10.1016/j.watres.2018.08.060 [61] SCHWERMER C U, KRZEMINSKI P, WENNBERG A C, et al. Removal of antibiotic resistant e. Coli in two norwegian wastewater treatment plants and by nano- and ultra-filtration processes[J]. Water Science and Technology, 2017, 77(4): 1115-1126. [62] ZHU Y J, WANG Y Y, ZHOU S, et al. Robust performance of a membrane bioreactor for removing antibiotic resistance genes exposed to antibiotics: Role of membrane foulants[J]. Water Research, 2018, 130: 139-150. doi: 10.1016/j.watres.2017.11.067 [63] NGUYEN T H, CHEN K L, ELIMELECH M. Adsorption kinetics and reversibility of linear plasmid DNA on silica surfaces: Influence of alkaline earth and transition metal ions[J]. Biomacromolecules, 2010, 11(5): 1225-1230. doi: 10.1021/bm901427n [64] SAEKI K, KUNITO T, SAKAI M. Effects of pH, ionic strength, and solutes on DNA adsorption by andosols[J]. Biology and Fertility of Soils, 2010, 46(5): 531-535. doi: 10.1007/s00374-010-0447-y [65] PEI M, ZHANG B, HE Y L, et al. State of the art of tertiary treatment technologies for controlling antibiotic resistance in wastewater treatment plants[J]. Environment International, 2019, 131: 105026. doi: 10.1016/j.envint.2019.105026 [66] CHENG H, HONG P Y. Removal of antibiotic-resistant bacteria and antibiotic resistance genes affected by varying degrees of fouling on anaerobic microfiltration membranes[J]. Environmental Science & Technology, 2017, 51(21): 12200-12209. [67] WANG K M, ZHOU L X, MENG S H, et al. Anaerobic membrane bioreactor for real antibiotic pharmaceutical wastewater treatment: Positive effect of fouling layer on antibiotics and antibiotic resistance genes removals[J]. Journal of Cleaner Production, 2023, 409: 137234. doi: 10.1016/j.jclepro.2023.137234 [68] KAYA Y, BACAKSIZ A M, BAYRAK H, et al. Investigation of membrane fouling in an anaerobic membrane bioreactor (AnMBR) treating pharmaceutical wastewater[J]. Journal of Water Process Engineering, 2019, 31: 100822. doi: 10.1016/j.jwpe.2019.100822 [69] CHEN P, XIE Q L, ADDY M, et al. Utilization of municipal solid and liquid wastes for bioenergy and bioproducts production[J]. Bioresource Technology, 2016, 215: 163-172. doi: 10.1016/j.biortech.2016.02.094 [70] SHIMADA T, LI X, ZILLES J L, et al. Effects of the antimicrobial tylosin on the microbial community structure of an anaerobic sequencing batch reactor[J]. Biotechnology and Bioengineering, 2011, 108(2): 296-305. doi: 10.1002/bit.22934 [71] WANG S J, HOU X C, SU H J. Exploration of the relationship between biogas production and microbial community under high salinity conditions[J]. Scientific Reports, 2017, 7: 1149. doi: 10.1038/s41598-017-01298-y [72] XIONG Y H, HARB M, HONG P Y. Performance and microbial community variations of anaerobic digesters under increasing tetracycline concentrations[J]. Applied Microbiology and Biotechnology, 2017, 101(13): 5505-5517. doi: 10.1007/s00253-017-8253-1 [73] BENERAGAMA N, LATEEF S A, IWASAKI M, et al. The combined effect of cefazolin and oxytertracycline on biogas production from thermophilic anaerobic digestion of dairy manure[J]. Bioresource Technology, 2013, 133: 23-30. doi: 10.1016/j.biortech.2013.01.032 [74] AYDIN S, INCE B, INCE O. Assessment of anaerobic bacterial diversity and its effects on anaerobic system stability and the occurrence of antibiotic resistance genes[J]. Bioresource Technology, 2016, 207: 332-338. doi: 10.1016/j.biortech.2016.01.080 [75] CETECIOGLU Z, INCE B, GROS M, et al. Chronic impact of tetracycline on the biodegradation of an organic substrate mixture under anaerobic conditions[J]. Water Research, 2013, 47(9): 2959-2969. doi: 10.1016/j.watres.2013.02.053 [76] PALA-OZKOK I, ORHON D. Chronic effect of erythromycin on substrate biodegradation kinetics of activated sludge[J]. Biochemical Engineering Journal, 2013, 81: 29-39. doi: 10.1016/j.bej.2013.10.002 [77] AMIN M M, ZILLES J L, GREINER J, et al. Influence of the antibiotic erythromycin on anaerobic treatment of a pharmaceutical wastewater[J]. Environental Science & Technology, 2006, 40(12): 3971-3977. [78] TANG L, FENG H D, LUAN X, et al. Occurrence, distribution, and behaviors of erythromycin a, production byproducts, transformation products, and resistance genes in a full-scale erythromycin a production wastewater treatment system[J]. Water Research, 2023, 245: 120640. doi: 10.1016/j.watres.2023.120640 [79] CETECIOGLU Z, INCE B, ORHON D, et al. Acute inhibitory impact of antimicrobials on acetoclastic methanogenic activity[J]. Bioresource Technology, 2012, 114: 109-1016. doi: 10.1016/j.biortech.2012.03.020 [80] HU J W, XU Q X, LI X M, et al. Sulfamethazine (SMZ) affects fermentative short-chain fatty acids production from waste activated sludge[J]. Science of the Total Environment, 2018, 639: 1471-1479. doi: 10.1016/j.scitotenv.2018.05.264 [81] LU M Q, NIU X J, LIU W, et al. Biogas generation in anaerobic wastewater treatment under tetracycline antibiotic pressure[J]. Scientific Reports, 2016, 6: 28336. doi: 10.1038/srep28336 [82] HE Y P, TIAN Z, LUAN X, et al. Recovery of biological wastewater treatment system inhibited by oxytetracycline: Rebound of functional bacterial population and the impact of adsorbed oxytetracycline on antibiotic resistance[J]. Chemical Engineering Journal, 2021, 418: 129364. doi: 10.1016/j.cej.2021.129364 [83] FENG H D, HU Y Q, TANG L, et al. New hydrolysis products of oxytetracycline and their contribution to hard cod in biological effluents of antibiotic production wastewater[J]. Chemical Engineering Journal, 2023, 471: 144409. doi: 10.1016/j.cej.2023.144409 [84] RUSANOWSKA P, HARNISZ M, ZIELIŃSKI M, et al. Individual and synergistic effects of metronidazole, amoxicillin, and ciprofloxacin on methane fermentation with sewage sludge[J]. CLEAN-Soil Air Water, 2020, 48(2): 1900281. doi: 10.1002/clen.201900281 [85] CZATZKOWSKA M, HARNISZ M, KORZENIEWSKA E, et al. The impact of antimicrobials on the efficiency of methane fermentation of sewage sludge, changes in microbial biodiversity and the spread of antibiotic resistance[J]. Journal of Hazardous Materials, 2021, 416: 125773. doi: 10.1016/j.jhazmat.2021.125773 [86] CHENG D L, NGO H H, GUO W S, et al. Improving sulfonamide antibiotics removal from swine wastewater by supplying a new pomelo peel derived biochar in an anaerobic membrane bioreactor[J]. Bioresource Technology, 2021, 319: 124160. doi: 10.1016/j.biortech.2020.124160 [87] WEI C H, SANCHEZ-HUERTA C, LEIKNES T, et al. Removal and biotransformation pathway of antibiotic sulfamethoxazole from municipal wastewater treatment by anaerobic membrane bioreactor[J]. Journal of Hazardous Materials, 2019, 380: 120894. doi: 10.1016/j.jhazmat.2019.120894 [88] DO T M, CHOI D, OH S, et al. Anaerobic membrane bioreactor performance with varying feed concentrations of ciprofloxacin[J]. Science of the Total Environment, 2022, 803: 150108. doi: 10.1016/j.scitotenv.2021.150108 [89] WANG X H, ZHANG J F, CHANG V W C, et al. Removal of cytostatic drugs from wastewater by an anaerobic osmotic membrane bioreactor[J]. Chemical Engineering Journal, 2018, 339: 153-161. doi: 10.1016/j.cej.2018.01.125 [90] SHEN L G, LEI Q, CHEN J R, et al. Membrane fouling in a submerged membrane bioreactor: Impacts of floc size[J]. Chemical Engineering Journal, 2015, 269: 328-334. doi: 10.1016/j.cej.2015.02.002 [91] AUBENNEAU M, TAHAR A, CASELLAS C, et al. Membrane bioreactor for pharmaceutically active compounds removal: Effects of carbamazepine on mixed microbial communities implied in the treatment[J]. Process Biochemistry, 2010, 45(11): 1826-1831. doi: 10.1016/j.procbio.2010.04.011 [92] JUNTAWANG C, RONGSAYAMANONT C, KHAN E. Entrapped cells-based-anaerobic membrane bioreactor treating domestic wastewater: Performances, fouling, and bacterial community structure[J]. Chemosphere, 2017, 187: 147-155. doi: 10.1016/j.chemosphere.2017.08.113 [93] LI B, YANG Y, MA L, et al. Metagenomic and network analysis reveal wide distribution and co-occurrence of environmental antibiotic resistance genes[J]. The ISME Journal, 2015, 9(11): 2490-502. doi: 10.1038/ismej.2015.59 [94] ZAREI-BAYGI A, HARB M, WANG P, et al. Microbial community and antibiotic resistance profiles of biomass and effluent are distinctly affected by antibiotic addition to an anaerobic membrane bioreactor[J]. Environmental Science:Water Research & Technology, 2020, 6(3): 724-736. [95] COSTA B F, ZAREI-BAYGI A, MD ISKANDER S, et al. Antibiotic resistance genes fate during food waste management-comparison between thermal treatment, hyperthermophilic composting, and anaerobic membrane bioreactor[J]. Bioresource Technology, 2023, 388: 129771. doi: 10.1016/j.biortech.2023.129771 [96] CUI T T, ZHANG S Y, YE J Y, et al. Distribution, dissemination and fate of antibiotic resistance genes during sewage sludge processing-a review[J]. Water Air & Soil Pollution, 2022, 233(4): 138. [97] World Health Organization. Technical brief on water, sanitation, hygiene and wastewater management to prevent infections and reduce the spread of antimicrobial resistance. Geneva: World health organization[DB/OL]. [2020-11-18]https://www.who.int/publications/i/item/9789240006416. [98] 聂宇, 陈娅婷, 孙照勇, 等. 污水/城市污泥中抗生素对厌氧消化的影响研究进展[J]. 应用与环境生物学报, 2020, 26(2): 479-488. [99] GóMEZ-PACHECO C V, SáNCHEZ-POLO M, RIVERA-UTRILLA J, et al. Tetracycline removal from waters by integrated technologies based on ozonation and biodegradation[J]. Chemical Engineering Journal, 2011, 178: 115-121. doi: 10.1016/j.cej.2011.10.023 [100] TANG M, LI F, YANG M, et al. Degradation of kanamycin from production wastewater with high-concentration organic matrices by hydrothermal treatment[J]. Journal of Environmental Sciences, 2020, 97: 11-18. doi: 10.1016/j.jes.2020.04.032 [101] YI Q Z, GAO Y X, ZHANG H F, et al. Establishment of a pretreatment method for tetracycline production wastewater using enhanced hydrolysis[J]. Chemical Engineering Journal, 2016, 300: 139-145. doi: 10.1016/j.cej.2016.04.120 [102] 张昱, 唐妹, 田哲, 等. 制药废水中抗生素的去除技术研究进展[J]. 环境工程学报, 2018, 12(1): 1-14. [103] HE Y P, TIAN Z, YI Q Z, et al. Impact of oxytetracycline on anaerobic wastewater treatment and mitigation using enhanced hydrolysis pretreatment[J]. Water Research, 2020, 187: 116408. doi: 10.1016/j.watres.2020.116408 [104] TIAN Y, TIAN Z, FENG H D, et al. Unveiling the threshold values of organic and oxytetracycline loadings for nitrification recovery of a full-scale pharmaceutical wastewater treatment system[J]. Chemical Engineering Journal, 2023, 463: 142487. doi: 10.1016/j.cej.2023.142487 [105] TIAN Y, TIAN Z, HE Y P, et al. Removal of denatured protein particles enhanced uasb treatment of oxytetracycline production wastewater[J]. Science of the Total Environment, 2022, 816: 151549. doi: 10.1016/j.scitotenv.2021.151549 [106] TANG M, GU Y, WEI D B, et al. Enhanced hydrolysis of fermentative antibiotics in production wastewater: Hydrolysis potential prediction and engineering application[J]. Chemical Engineering Journal, 2020, 391: 123626. doi: 10.1016/j.cej.2019.123626 [107] 田野. 基于强化水解预处理的土霉素生产废水处理工艺研究[D]. 北京: 中国科学院生态环境研究中心, 2023. [108] WU D N, DAI S T, FENG H D, et al. Persistence and potential risks of tetracyclines and their transformation products in two typical different animal manure composting treatments[J]. Environmental Pollution, 2024, 341: 122904. doi: 10.1016/j.envpol.2023.122904 [109] DUNCAN J, BOKHARY A, FATEHI P, et al. Thermophilic membrane bioreactors: A review[J]. Bioresource Technology, 2017, 243: 1180-1193. doi: 10.1016/j.biortech.2017.07.059 [110] JANG H M, SHIN J, CHOI S, et al. Fate of antibiotic resistance genes in mesophilic and thermophilic anaerobic digestion of chemically enhanced primary treatment (CEPT) sludge[J]. Bioresource Technology, 2017, 244: 433-444. doi: 10.1016/j.biortech.2017.07.153 [111] 马清佳, 田哲, 员建, 等. 9种抗生素对污泥高温厌氧消化的急性抑制[J]. 环境工程学报, 2018, 12(7): 2084-2093. [112] TIAN Z, CHI Y Z, YU B, et al. Thermophilic anaerobic digestion reduces args in excess sludge even under high oxytetracycline concentrations[J]. Chemosphere, 2019, 222: 305-313. doi: 10.1016/j.chemosphere.2019.01.139 [113] ASLAM M, CHARFI A, LESAGE G, et al. Membrane bioreactors for wastewater treatment: A review of mechanical cleaning by scouring agents to control membrane fouling[J]. Chemical Engineering Journal, 2017, 307: 897-913. doi: 10.1016/j.cej.2016.08.144 [114] NGUYEN T T, BUI X T, LUU V P, et al. Removal of antibiotics in sponge membrane bioreactors treating hospital wastewater: Comparison between hollow fiber and flat sheet membrane systems[J]. Bioresource Technology, 2017, 240: 42-49. doi: 10.1016/j.biortech.2017.02.118 [115] ZHANG W X, TANG B, BIN L Y. Research progress in biofilm-membrane bioreactor: A critical review[J]. Industrial & Engineering Chemistry Research, 2017, 56(24): 6900-6909. [116] LI Z H, YUAN L, YANG C W, et al. Anaerobic electrochemical membrane bioreactor effectively mitigates antibiotic resistance genes proliferation under high antibiotic selection pressure[J]. Environment International, 2022, 166: 107381. doi: 10.1016/j.envint.2022.107381 [117] LI Z H, YUAN L, GENG Y K, et al. Evaluating the effect of gradient applied voltages on antibiotic resistance genes proliferation and biogas production in anaerobic electrochemical membrane bioreactor[J]. Journal of Hazardous Materials, 2021, 416: 125865. doi: 10.1016/j.jhazmat.2021.125865 [118] DING A Q, FAN Q, CHENG R, et al. Impacts of applied voltage on microbial electrolysis cell-anaerobic membrane bioreactor (MEC-AnMBR) and its membrane fouling mitigation mechanism[J]. Chemical Engineering Journal, 2018, 333: 630-635. doi: 10.1016/j.cej.2017.09.190 -


 下载:
下载:

